10月16日,南京中医药大学医学院﹒整合医学学院病原生物学系林炜教授课题组在国际知名学术期刊Nucleic Acids Research(中科院生物一区)在线发表了题为“Structural basis of three different transcription activation strategies adopted by a single regulator SoxS”的研究论文,首次揭示了细菌AraC家族全局性转录调控因子SoxS对革兰氏阴性病原菌耐药基因、氧化还原压力应激基因等的转录激活调控机制,为抗耐药菌感染药物的研发提供了新型靶点和研究思路。该工作是林炜教授团队继Mol. Cell(2017, 2018)、Nat Commun(2019)、Nucleic Acids Res(2020, 2021, 2022) 之后在病原菌转录调控和新型抗耐药菌药物靶点发现领域取得的又一项全新研究成果。

革兰阴性病原菌是引起人类感染性疾病的重要病原菌,因临床常用抗菌药物种类少,细菌耐药性问题日益严峻,其中,鲍曼不动杆菌、肠杆菌等革兰阴性病原菌已被世界卫生组织列为当前急需开发新型抗生素的重点耐药性细菌。研究表明,外膜孔道蛋白通透性改变、主动外排泵系统等活性增强等是导致革兰阴性病原菌多重耐药的重要机制,而外膜孔道蛋白以及外排泵系统的表达水平受到多种转录因子的协同调控,进而降低细胞内药物浓度导致细菌耐药,细菌AraC家族中的SoxS、Rob等蛋白即为典型代表,与细菌的耐药性、应激压力响应、毒力等紧密相关(图1A)。
与该团队近期报道(Nucleic Acids Res,2022)的Rob蛋白不同,SoxS是AraC家族蛋白中最小的成员,属于全局性调控因子,可通过与RNA聚合酶作用使膜孔蛋白基因表达下调、多重耐药操纵子表达上调,从而导致细菌耐药性增强。另外,SoxS可以调控多种不同类型的启动子DNA(含多种耐药基因启动子),而且能分别以“正向”和“反向”结合的方式激活基因转录。如此小的蛋白是如何灵活调控不同结构特征的启动子起始转录的呢?目前仍认识不够清楚。
通过采用冷冻电镜技术,我校林炜教授团队成功解析了含大肠杆菌RNA聚合酶、SoxS蛋白以及不同启动子DNA的转录激活复合物SoxS-TAC的结构,成功捕获了SoxS单体灵活调控不同类型启动子的功能状态。结构分析表明SoxS除通过保守的HTH motif识别启动子DNA的SoxS结合元件(soxsbox)外,还可以与RNA聚合酶的不同保守结构域相互作用促进转录起始(图1B)。与SoxS-TACI、SoxS-TACII两类复合物不同,在SoxS-TACIII中,soxsbox距离RNA聚合酶更远而且方向相反,巧妙的是,SoxS能以“反向”结合的方式识别soxsbox,并借助于多种复杂相互作用成功稳定复合物并促进转录起始。该研究系统阐明了SoxS单体灵活调控细菌耐药基因等多种启动子的分子机制,可为新型抗耐药菌感染药物的研发提供新的靶点和研究思路。同时,该研究首次呈现并揭示了细菌RNA聚合酶的2个αCTD在基因转录调控方面的普遍调控意义,可为合成生物学家高效调控细菌基因表达提供重要元件。
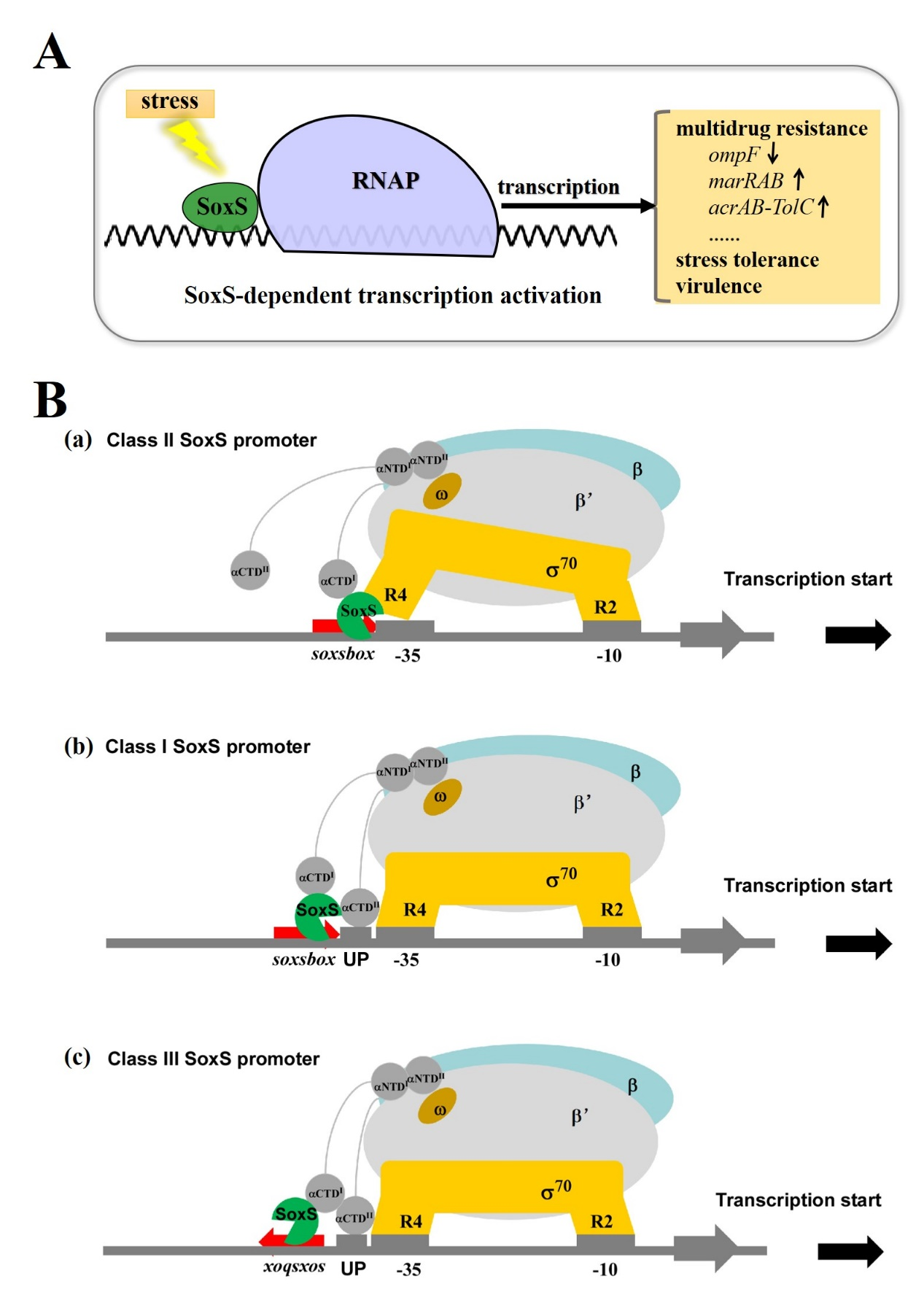
图1 SoxS蛋白转录激活调控的生理意义与模型
南京中医药大学为该论文的第一作者和通讯作者单位,林炜教授为该论文的第一通讯作者;史婧副教授为该论文的第一作者和共同通讯作者;博士研究生王福林、硕士研究生王璐为该论文的共同第一作者。

